- سلولی و مولکولی
- 01. دوره جامع آزمایشگاه ژنتیک
- 02. دوره جامع کارآموزی مولکولی
- 03. دوره کارآموزی ارشد آزمایشگر
- 47. دوره آنالیز داده های NGS
- 05. دوره کارآموزی جامع کاربری دستگاه فلوسایتومتری
- 16. دوره طراحی پرایمر و اصول PCR
- 11. دوره کاربری دستگاه Real time PCR
- 46. دوره پژوهشگر شو
- 21. کارگاه QF-PCR
- 04.دوره کارآموزی جامع تکنسین آزمایشگاه ژنتیک (کاریوتایپ)
- 13. دوره جامع ارشد مهندسی ژنتیک (کلونینگ) و دوره جامع کشت سلول (رده سلول سرطانی)
- 08. دوره کشت سلول (رده سلول سرطانی)
- 15. کارگاه SDS-PAGE و وسترن بلات
- 17. دوره آنالیز کروموزوم های انسانی (مقدماتی و پیشرفته)
- 31. کارگاه معرفی تکنولوژی های ویرایش ژنوم یوکاریوتی با تاکید بر تکنولوژی مدرن CRISPR/Cas9
- میکروبیولوژی
- 09. دوره جامع ارشد مهندسی ژنتیک (کلونینگ)
- 20. کارگاه بیان، استخراج و تخلیص پروتئین از میزبان باکتریایی
- 23. نرم افزار های مولکولی
- 55. دوره کاربری دستگاه فرمانتور
- 56. دوره میکروب شناسی آزمایشگاه
- 57. ارزیابی بیان پروتئین نوترکیب
- 58. کارگاه تولید پروتئین تک سلولی
- 59. کارگاه آنالیز متابولیت های ثانویه در گیاهان دارویی
- نانو فناوری
- 45. دوره الکتروریسی
- 34. تولید نانو ذرات به روش میکروامولسیون
- 35. تولید نانو ذرات به روش آسیاب گلوله ای
- 36. تولید نانو ذرات به روش سل ژل
- 37. تولید نانو ذرات به روش هیدروترمال
- 38. متصدی تولید نانو لوله های کربنی با روش CVD
- 39. متصدی توليد نانو كامپوزيت های پايه پليمری گرما نرم
- 60. آزمایشگر ارشد کروماتوگرافی گازی
- زیست پزشکی
- 06. دوره مهندسی بافت
- 14. دوره ایمونوتراپی سرطان
- 18. کارگاه حیوانات آزمایشگاهی (موش و رات)
- 19. کارگاه آنالیز داده های زیستی
- 48. کارگاه کاربری دستگاه الایزا
- 49. کارگاه جامع ساخت هیدروژل ها (تمام عملی)
- 50. کارگاه مهندسی حاملهای دارو رسان
- 51. کارگاه جامع زیستسازگاری و آزمونهای بیولوژیک
- 64. دوره ایمونوهیستوشیمی
- 65. دوره تکنیک های نوین علوم زیستی
- 66. کارگاه واکسن های نوترکیب
- آموزشی پژوهشی
- دوره های عمومی
- 07. دوره جامع کارآموزی در آزمایشگاه (پذیرش، نمونه گیری، تزریقات با سرنگ و ونوجکت، نسخه خوانی)
- 10. آموزش کنترل کیفی، استانداردسازی و مستندسازی آزمایشگاههای تشخیص پزشکی
- 18. کارگاه حیوانات آزمایشگاهی (موش و رات)
- 48. کارگاه کاربری دستگاه الایزا
- 32. دوره آنلاین WGCNA
- 61. دوره کاربر پایگاه های علوم زیستی
- 63. تحلیلگر آزمايشات علوم زيستی و بیوتکنولوژی با نرم افزار Minitab
- برنامه نویسی
- 25. دوره کامل مجازی آموزش برنامه نویسی پایتون (مقدماتی تا پیشرفته)
- 26. دوره آفلاین جامع برنامه نویسی R (مقدماتی و پیشرفته)
- 27. دوره آفلاین جامع برنامه نویسی پیشرفته R سطح ۱(TCGA)
- 28. دوره آفلاین برنامه نویسی پیشرفته R سطح 2: functional Enrichmet و نمودارها در R
- 29. دوره آفلاین برنامه نویسی پیشرفته R (دوره ی پیشرفته ی 3: GEO)
- 30. دوره آفلاین ceRNA
32. دوره آنلاین WGCNA
آموزشگاه آزاد نانو زیست فناوری اوژن اولین آموزشگاه رسمی فنی و حرفه ای در رشته نانو زیست فناوری در تهران برگزار میکند:
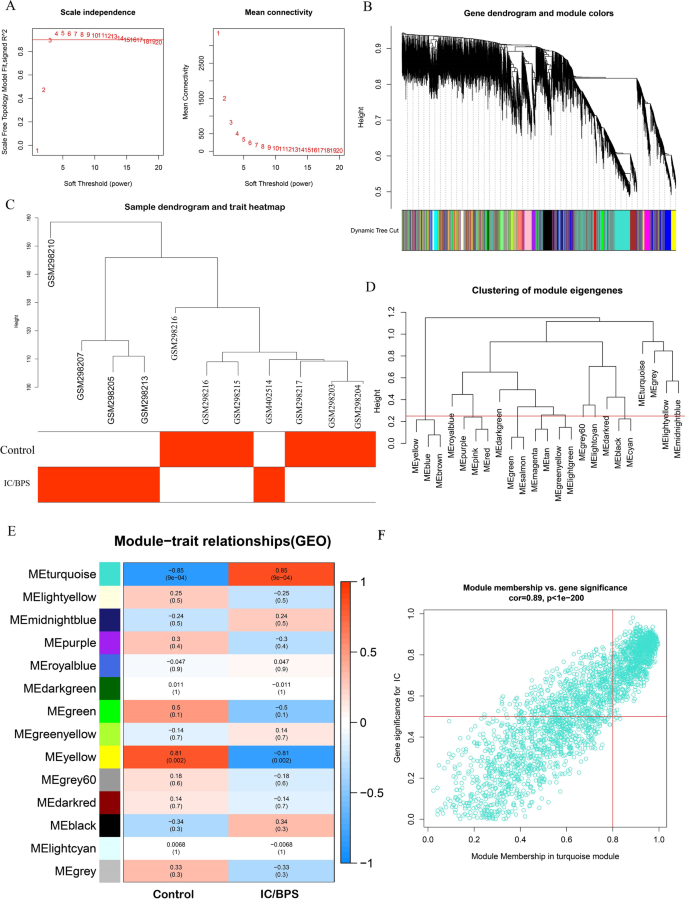
دوره WGCNA (آنلاین)
سرفصل و محتوا :
- مباحث تئوری شبکه ها و WGCNA
- شروع کدنویسی ها با پکیج WGCNA
- کلاسترینگ (sample cluster)
- درخت کلاسترینگ (sample tree)
- شناسایی نمونه ی outlier (outlier identifier)
- ارتباط نمونه ها با دیتای کلینیکال (clinical trait-sample dendrogram)
- شناسایی ماژول ها (module detection)
- شناسایی عدد پاور (power soft) و مبحث scale free
- تشکیل ماتریکس TOM
- Merge کردن ماژول ها
- ایجاد ماژول اینگژن (eigenegene)
- ارتباط ماژول-کلینیکال (module-clinical trait)
- هیپ مپ همبستگی (correlation heatmap)
- Gene Significance and Module Membership
- شناسای هاب ماژول ها
- شناسایی هاب ژن ها
- Annotation کردن ژن ها
🔴قیمت استثنایی
🔴ظرفیت محدود
پیشرفت هریک از شما پیشرفت ماست
❖ ── ✦ ──『✙ – ✙』── ✦ ── ❖
برای اطلاع از جزئیات ، ثبت نام و مشاوره می توانید از طریق شماره تلفن، واتساپ، اینستاگرام ، تلگرام و وبسایت آموزشگاه آزاد نانو زیست فناوری اوژن اقدام کنید.
www.ogene-tech.com
tel: 021-44961487-09120169816
whats app: 09233093463
t.me/Ogenetechnology
instagram.com/ogenetech
tel: 021-44961487-09120169816
whats app: 09233093463
t.me/Ogenetechnology
instagram.com/ogenetech
«مهارت شما آینده شماست»
Description
Description
دوره آنلاین WGCNA
شبکه همبیانی ژنها (به انگلیسی: Gene Co-Expression Network) یک گراف بدون جهت است. در این گراف هر رأس معادل با یک ژن است. همچنین بین دو راس یال وجود دارد، اگر و تنها اگر رابطه همبیانی معناداری میان ژنهای متناظر آن دو رأس موجود باشد. در اینجا منظور از همبیانی این است که الگوی بیان دو ژن، به معنای کم یا زیاد شدن بیان آنها یکسان باشد. شبکههای همبیانی ژنها به کمک مجموعه دادهای از بیان تعدادی ژن در نمونههای مختلف بدست میآید. از آنجا که شبکه همبیانی ژنها یک گراف بدون جهت است، یک روش مناسب برای نمایش آن ماتریس همسایگی است. در این روش اگر همبیانی دو ژن از آستانهای مشخص بالاتر باشد بین آنها یال متصور میشویم و درایههای ماتریس همسایگی صفر یا یک هستند.
همچنین میتوان مدلی پیچیدهتر برای شبکه همبیانی ژنها متصور شد که در آن یالها وزنی در بازه [0,1] داشته باشند. در این روش به اصطلاح از آستانهای نرم استفاده میشود. در مقابل این روش به روش قبلی که یالها وزن نداشتند، استفاده از آستانه سخت گفته میشود. در روش آستانه سخت ممکن است یک مقدار اختلاف کمی با آستانه داشته باشد و از آن کمتر باشد، در این صورت یال مربوط به این مقدار ترسیم نمیشود که از معایب این روش محسوب میشود. همچنین از نظر زیستی معنیدارتر است که رابطه همبیانی دو ژن را با یکوزن مشخص کنیم. البته باید توجه داشت در مقابل دقت بیشتری که در شبکههای وزندار بدست میآید، تحلیل گراف حاصل سختتر میشود.
شبکههای همبیانی ژنها کاربردهای متعددی دارند. برای نمونه این شبکهها میتوانند ابزاری مناسب در شناختن ژنهایی باشند که کارکرد زیستی آنان چندان مشخص شده نیست. همچنین این شبکهها میتوانند در تحلیل ارتباط مکانیزمهای مولکولی و فرایندهای زیستی مفید واقع شوند.
در حال حاضر شبکههای بسیاری توسط دادههای حاصل از ریزآرایه دیانای تولید شدهاند.
ساختن شبکه همبیانی ژنها
ساخت شبکههای همبیانی ژنها به کمک دادهٔ بیان مجموعهای از ژنها در تعدادی نمونه انجام میشود. برای این کار ابتدا بیان مجموعهای از ژنها را در نمونههای مختلف اندازهگیری میشود. اگر داده نیاز به هرگونه نرمالسازی داشته باشد، در این مرحله انجام میشود. اطلاعات بیان ژن در نمونههای مختلف را میتوان در یک ماتریس که ستونهای آن متناظر با نمونهها و سطرهای آن متناظر با ژنهاست، نمایش داد. سپس به کمک دادهٔ بدست آمده، امتیازی تحت عنوان امتیاز شباهت میان هر دو ژن محاسبه میشود. این امتیازها را میتوان در یک ماتریس مربعی، که سطرها و ستونهای آن متناظر با ژنها هستند، ذخیره کرد. سپس برای هر دو ژنی که امتیاز شباهت آنها از یک آستانه مشخص بیشتر بود، یک یال در گراف متناظر قرار داده میشود. این کار را میتوان با اعمال یک تبدیل ساده بر روی ماتریس شباهت ژنها انجام داد. برای بدست آوردن ماتریس همسایگی گراف کافی است در ماتریس شباهت ژنها، درایههای بزرگتر مساوی از آستانه را به یک و بقیه درایهها به صفر تبدیل شوند. برای ساختن شبکه همبیانی ژنها باید روش محاسبه امتیاز شباهت و محاسبه آستانه مشخص شوند.

محاسبه امتیاز شباهت
میزان بیان یک ژن در نمونههای مختلف را میتوان به صورت یک بردار نمایش داد. پس برای محاسبه امتیاز شباهت دو ژن، باید امتیازی را بین دو بردار بیان ژن محاسبه کنیم. برای محاسبه این امتیاز میتوان از روشهایی مانند ضریب همبستگی پیرسون، ضریب همبستگی رتبهای اسپیرمن و اطلاعات متقابل استفاده کرد که امتیاز حاصل از تمام این روشها در بازه [0,1] یا [−1,1] قرار دارد. در برخی موارد نیز امتیازهایی محاسبه میشوند که این ویژگی را ندارند، یک نمونه از چنین امتیازهایی فاصله اقلیدسی است. در برخی از موارد نیز از روشهایی بر مبنای همبستگی جزئی مانند مدلهای گاوسی گرافیکی استفاده میشود.
محاسبه آستانه
یکی از روشهای ساده محاسبه آستانه ثابت گرفتن اندازه شبکه (تعداد یالهای گراف متناظر) است. در این روش تعداد یالها معلوم است و امتیاز تمام جفت ژنها نیز معلوم است. پس آستانه به سادگی مشخص میشود. در روش دیگر به جای تعیین آستانه برای امتیاز شباهت، آستانهای برای سطح معنادار بودن امتیاز تعیین میشود. در این روشها ابتدا برای هر کدام از امتیازهای شباهت یک پی-مقدار محاسبه میشود. سپس با توجه به پی-مقدار و سطح معنادار بودن تعیین شده یالها را رسم میکنند. برای محاسبه پی-مقدار معمولاً از آزمون جایگشتی یا آزمون زد استفاده میشود.
استفاده از آستانهٔ نرم
در این روش هم ابتدا نیاز داریم، ماتریس امتیاز شباهتها را محاسبه کنیم. در این قسمت فرض میکنیم که امتیاز شباهت در بازهای مانند [0,1] یا [−1,1] قرار دارد. برای نمونه میتوانیم برای محاسبه امتیاز از ضریب همبستگی پیرسون استفاده کنیم. امتیاز شباهت و وزن یال میان ژن 



استفاده از تابع سیگموئید
در این روش از تابع سیگموئید به همراه دو پارامتر 





استفاده از تابع توانی
در این روش یک پارامتر 

و …
Reviews (0)
Be the first to review “32. دوره آنلاین WGCNA” لغو پاسخ



















Reviews
There are no reviews yet.